Hash-Tabelle
(engl. hash table od. hash map)
Hash-Tabelle
- Wir führen eine Liste mit einer fixen Anzahl an
-
Eine Hashfunktion muss bestimmt werden, die unsere Daten in den Bereich
-
Daten können dann in den so bestimmten Slot eingefügt werden
-
z.B.
-
Es lässt sich jetzt auch ein Füllfaktor
-
Anwendungsfall: Wir können speichern ob ein Element in einer Liste vorhanden ist oder nicht
Hash-Tabelle
- In der Praxis wird eine Hashfunktion für jeden Datentyp benötigt
- Normalerweise implementieren wir diese nicht selbst
Beispiel einer Hashfunktion - my_hash_functions.py
- Verwendet die Modulo-Operation als rudimentäre Hashfunktion
def my_hash_fun(item, m):
#use modulo as rudimentary hash function
return item % m
a_list = [1, 16, 8, 4, 25, 94, 46]
print([my_hash_fun(x, 11) for x in a_list])
# [1, 5, 8, 4, 3, 6, 2]
- In weiter Folge ist
 Beispiel einer Hashfunktion für Strings
Beispiel einer Hashfunktion für Strings
- Exemplarisch ist hier eine Hashfunktion für Strings gegeben
def my_str_hash_fun(a_str, m):
#ord(...) returns the unicode value of a character
return sum([ord(x) * (i + 1) for i, x in enumerate(a_str)]) % m
a_string = "Hello World!"
print(my_str_hash_fun(a_string, 11)) #> 1
Hash-Tabelle
-
Suche in unserer Hash-Tabelle jetzt durch Anwenden der Hashfunktion auf die Daten und überprüfen, ob der Slot ein Element enthält →
→ Wir suchen den Wert 46 → -
Was würde aber passieren, wenn wir noch das Element
70in die Hash-Tabelle einfügen wollen?
4→ keine eindeutige Zuweisung mehr möglich
Hash-Tabelle
Lösung für hash collisions
- Verwendung einer perfekten Hashfunktion → nur selten möglich
- Wenn der vom Hash bestimmte Slot bereits besetzt ist wird der nächste freie gewählt → offene Adressierung mit linearem Sondieren
- Dieser Prozess muss auch bei allen Operationen berücksichtigt werden
- Entsprechend werden Hash-Tabellen mit hohem Füllfaktor
Assoziatives Array
Assoziatives Array mit Schlüssel-Wert-Paaren
- Eine Hash-Tabelle speichert prinzipiell nur Werte
- In der Praxis wollen wir aber meist ein
Tupelaus einer Bezeichnung und einem dazugehörigen Wert speichern - Üblicherweise wird deshalb von einem Schlüssel-Wert-Paar oder key-value pair gesprochen
- Der Schlüssel wird mit der Hashfunktion in einen Index umgewandelt → der Wert wird dann in diesem Slot abgelegt*
* Ganz strikt betrachtet ist das assoziative Array der abstrakte Datentyp & die Hash-Tabelle eine mögliche Implementierung.
Assoziatives Array mit Schlüssel-Wert-Paaren
- In Python bildet ein Dictionary (
dict) ein assoziatives Array ab
Python Dictionary - dictionary_example.ipynb
- Schlüssel-Wert-Paare:
- Schlüssel: unveränderlich (immutable) & eindeutig → z.B. Strings, Zahlen, Tupel
- Wert: beliebig & müssen nicht eindeutig sein
my_dict = {
"key1": "value1",
"key2": 2,
3: ("value", 3),
}
print(f"{my_dict["key1"]} | {my_dict["key2"]} | {my_dict[3]}")
# value1 | 2 | ("value", 3)


**kwargs in Python
- Optionale Schlüsselwortargumente für Funktionen/Methoden werden auch als
dictübergeben
→ (siehe 01_03_Python_Grundlagen_Funktionen_und_Module)
Assoziatives Array mit Schlüssel-Wert-Paaren
- Wenn wir wieder unsere Tabelle der Zeitkomplexitäten betrachten, können wir sehen, dass suchen, einfügen & entfernen von Elementen in einem assoziativen Array mit
Hinweis: "Access" bezeichnet hier den direkten Zugriff über einen Index → für eine Hash-Tabelle nicht vorhanden
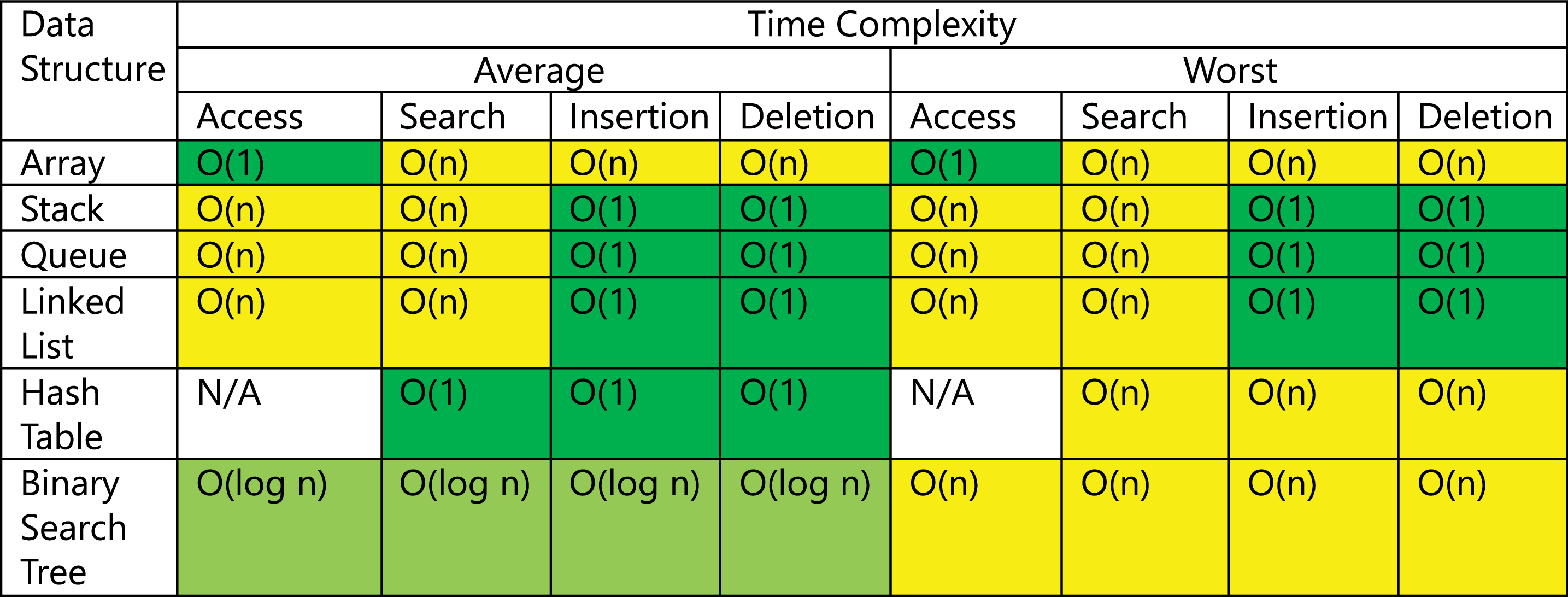
Bildquelle: [Althoff 2021]
 Aufgabe
Aufgabe
- Wir betrachten nochmals das Beispiel des Reverse-Complement einer DNA-Sequenz → Git-Repository
- Überarbeiten Sie Ihre Lösung für
reverse_complement(dna_sequence)mit den beiden Listen so, dass Sie ein Dictionary verwenden - Welche Vorteile bietet dieser Ansatz gegenüber jenem mit Listen?
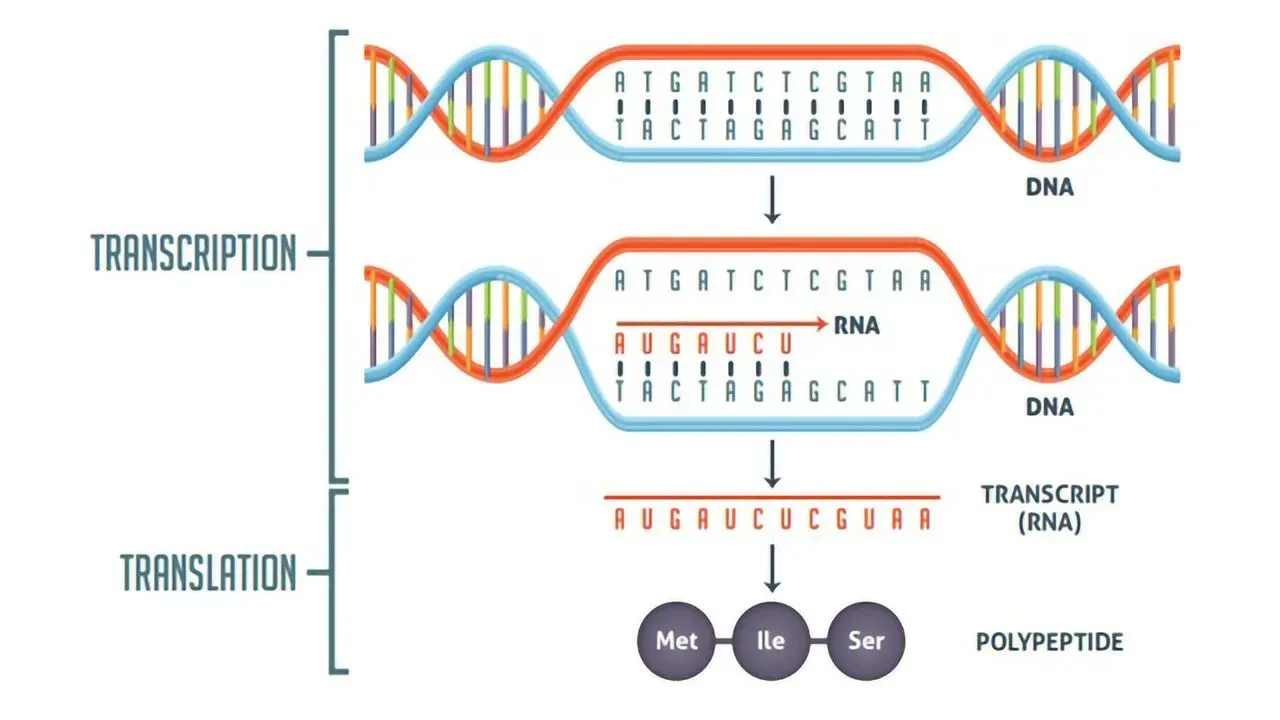
Beispiel Reverse-Complement-Übersetzer mit Dictionary
- Liste könnten sich zur Laufzeit verändern → Alphabete passen nicht mehr
- Listen sind hier ineffizient:
- Für jedes zu übersetzende Nukleotid muss der Index in der ersten Liste gefunden werden
- Erst dann kann der Index in der zweiten Liste genutzt werden
- In Python sind Listen als singly linked list implementiert → wir müssen durch die Liste iterieren um den passenden Index zu finden
- Dictionaries lösen beide Probleme gleichzeitig
Beispiel Reverse-Complement-Übersetzer mit Dictionary
Beispiel Reverse-Complement-Übersetzer mit Dictionary
Implementierung - reverse_complement.py
def reverse_complement_with_dict(dna_sequence):
complement_dict = {'A': 'T',
'T': 'A',
'G': 'C',
'C': 'G'}
reverse_comp_sequence = []
for nucleotide in dna_sequence:
if nucleotide in complement_dict:
reverse_comp_sequence.append(complement_dict[nucleotide])
else:
# In case of non-standard characters
reverse_comp_sequence.append("_")
reverse_comp_sequence.reverse()
return "".join(reverse_comp_sequence)
Unittests - ident zu vorherigem Beispiel
import unittest
from reverse_complement import reverse_complement_with_dict
class TestReverseComplement(unittest.TestCase):
def test_reverse_complement(self):
dna_sequence = "ATGATCTCGTAA"
reverse_complement_sequence = reverse_complement_with_dict(dna_sequence)
self.assertEqual(reverse_complement_sequence, "TTACGAGATCAT")
Serialisierung von Datenstrukturen
Serialisierung von Datenstrukturen
- Unter Serialisierung verstehen wir die Umwandlung von Datenstrukturen in ein Format, das gespeichert oder übertragen werden kann (z.B. als ASCII-codierte Bitfolge)
- Die nicht unbedingt gemeinsam im Arbeitsspeicher liegenden Datenstrukturen können so in eine serielle Speicher und übertragbare Form gebracht werden
- Ein Standard hierfür ist JSON (JavaScript Object Notation) → menschenlesbar und maschinenunabhängig
 Aufgabe
Aufgabe
- Wir wollen unsere Sensorklasse aus
dictionary_example.ipynberweitern - Unsere Sensoren bekommen ein Kalibrierungsdatum → wir wollen nun in der Lage sein unsere Sensoren anhand dieses Merkmales zu sortieren
- Wir wollen unsere Sensoren auch serialisieren können → wir nutzen JSON als Format
- Eine serialisierte Datei sollte auch immer wieder eingelesen werden können
Beispiel Sensorklasse
Musterlösung - sensor_with_calibration.ipynb
- Wir erweitern unsere Sensorklasse um ein Kalibrierungsdatum:
datetime - Wir müssen die Dunder-Methode
__lt__(less than) implementieren um unsere Sensoren vergleichen zu können - Wir nutzen das
json-Package um unseren Sensor zu serialisieren und als JSON-Datei zu speichern
 Hausübung
Hausübung
- Vergleiche der Worthäufigkeit in Texten
- Motivierte Studierende könne hier auch noch Texte anderer Autoren hinzufügen und die Worthäufigkeit zwischen Autoren vergleichen
- Dies kann genutzt werden um zu überprüfen ob zwei Texte vom selben Autor stammen